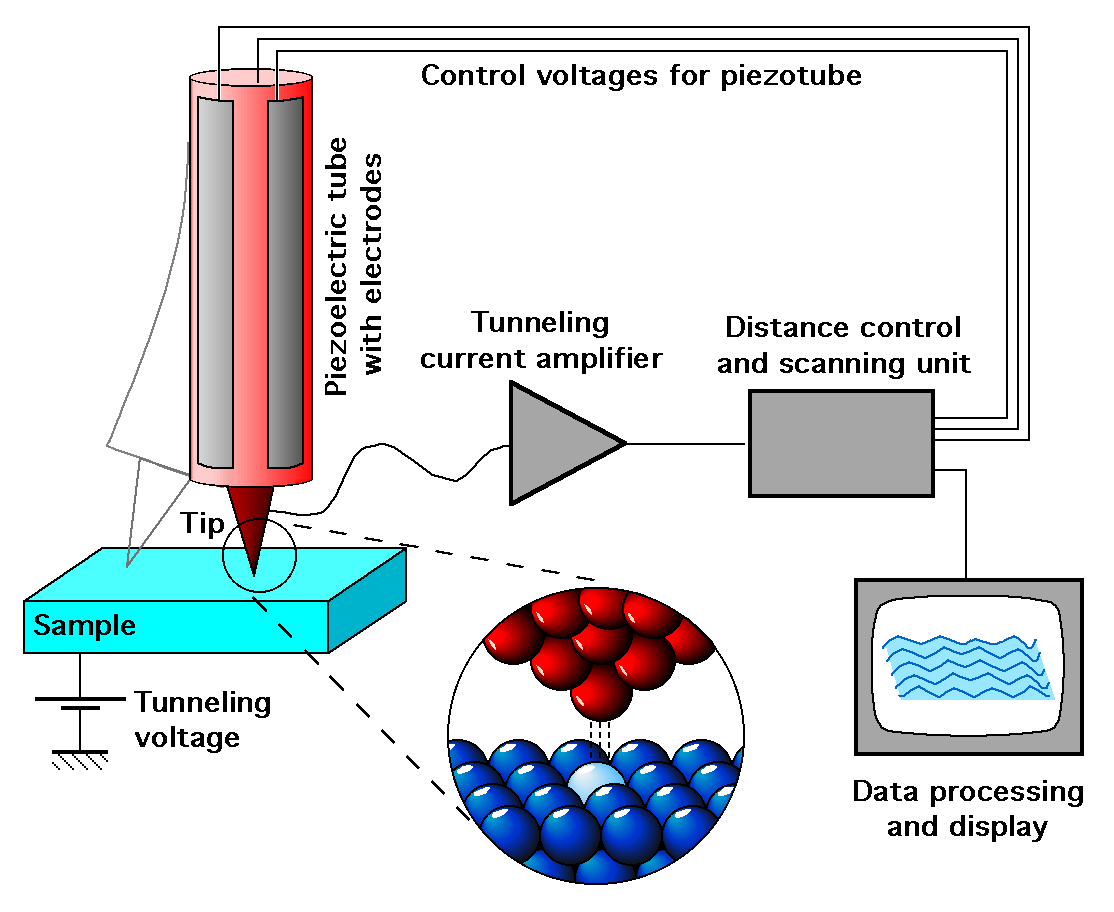
Kurs ten obejmował dwie techniki SPM: Scanning Tunneling Microscopy i Scanning Force Microscopy i został podzielony na trzy części. Część pierwsza obejmowała omówienie podstawowych składników STM i SFM z omówienie szczegółowo potencjalnych problemów na jakie można się natknąć przy pomiarach i jak je ewentualnie rozwiązywać. Dodatkowo podane zostały podstawy teoretyczne wraz z modelami matematycznymi.
Druga część to szczegółowe omówienie SFM na podstawie dwóch przeglądowych artykułów:
- Advances in atomic force microscopy, Franz J. Giessibl
- Atomic force microscopy and spectroscopy, Yongho Seo, Wonho Jhe
Oprócz tego omówiono różne modyfikacje SFM, t.j.: MFM – MRFM – EFM – KPFM –
LFM or FFM – AFAM – CAFM – TUNA.
Ciekawym pomysłem jest spięcie SFM z MRI. W artykule jaki został nam przedstawiony, osiągnięto rozdzielczość 90 nm
[1][
2]. SFM wraz z ESR daje również możliwość obserwacji pojedynczych spinów [
3] z niezwykłą precyzją. Warto przeczytać ten artykuł. Szczególnie interesujący jest sam sposób połączenia oscylacji dźwigni (
cantilever) z oscylacją pola elektromagnetycznego.
Ostatnia część to kilka godzin zajęć praktycznych w laboratorium, gdzie można było zobaczyć jak się takie pomiary przeprowadza, jak przygotowuję się próbki etc.
Egzamin jak zwykle ustny, po jednym z każdej części.